Система одновременного ГМО-скрининга
Создание генетически модифицированных культур являлось ответом на продовольственный вопрос постоянно растущей численности населения планеты. Благодаря новым биотехнологическим подходам растения приобрели такие сельскохозяйственно важные качества, как устойчивость к гербицидам, устойчивость к насекомым и устойчивость к абиотическим стрессам [1].
Первым генетически модифицированным организмом (ГМО), одобренным для коммерческого использования, был томат Flavr-Savr, выпущенный на рынок в 1994 году. В геноме данного томата была ингибирована выработка фермента полигалактуроназы, который ответственен за расщепление пектина в клеточной стенке плодов. Таким образом, замедлялось размягчение плодов после созревания и возрастала их лежкость.
Однако в 1998 году, после так называемого дела Арпада Пуштаи, произошел фундаментальный сдвиг общественных настроений в сфере потребления ГМО.
Это привело к формированию противоречивого отношения к ГМО-продуктам во всем мире и ужесточению законодательного контроля над ними.
На данный момент в мире зарегистрировано 11 трансгенных томатов, генетические модификации которых направлены на модуляцию выработки этилена, устойчивость к чешуекрылым и устойчивость к вирусу огуречной мозаики [3].
И хотя среди 28 ГМ-культур, зарегистрированных в нашей стране, нет ни одного томата, всегда существует вероятность случайного попадания такого продукта на отечественный рынок.
В рамках контроля ГМ-растений и потребительского «права знать» в лабораториях селекционно-семеноводческой компании «Гавриш» проводится обширная сертификация поступающих в коллекцию семенных образцов исходного селекционного материала, где наравне с анализами на наличие латентных фитопатогенов проводится анализ на наличие трансгенных конструкций, так как очень важно понимать, с чем мы работаем и какой генотип будет привнесен в селекционный процесс.
Для данного анализа мы используем только передовые решения в области прямого метода диагностики ГМО, основанного на анализе ДНК. Говоря другими словами, мы подвергаем все партии поступающих в коллекцию семян real-time PCR-анализу. В этом исследовании была разработана диагностическая система из пяти мишеней, основанная на SYBR®Green, позволяющих обнаруживать последовательности ДНК nptII, cmvCP, p35S, tNOS и PacPAP.
Для оценки совместной ПЦР-реакции всех пяти маркеров была синтезирована искусственная матрица, содержащая сайт отжига для каждой пары праймеров. Специфичность методов была проверена на ряде видов товарных культур и на контрольных ГМО-образцах.

Концепция диагностических систем
Для сертификации семенной продукции используется качественный анализ ГМ-конструкций.
Исходя из данного критерия, ученые компании «Гавриш» создали собственные маркерные системы на основе кривых плавления с интерколирующим красителем SYBR®Green. Выбор данного метода является оправданным с точки зрения поточности анализа и его экономической эффективности.
Также данный метод особенно удобен в тех случаях, когда главной задачей анализа является детекция ГМ-конструкции без необходимости точной дифференциации чужеродного фрагмента ДНК.
Для выбора потенциальных мишеней наши ученые проанализировали список из 11 зарегистрированных трансгенных томатов и возможных ГМ-конструкций, которые теоретически могут встречаться в огурце. В результате было выбрано пять ДНК мишеней, приведенных в таблице 1.

Первой мишенью является неомицин-фосфотрансфераза II (nptII) [5]. Данный ген используется в ГМ-конструкциях как маркер отбора, обусловливающий устойчивость к аминогликозидным антибиотикам.
Второй мишенью является белок капсидной оболочки вируса огуречной мозаики (CMV cp) [6]. Данный ген обусловливает постоянную устойчивость томата к вирусу огуречной мозаики.
Третьей мишенью является 35S промотор вируса мозаики цветной капусты (CaMV 35s) [7]. Данный ген входит в стандартные перечни всех базовых систем для определения наличия/отсутствия ГМ-структур в анализируемом геноме.
Четвертой мишенью является ген нопалин-синтазы из Agrobacterium tumefaciens (tNOS) [8]. Как и 35s, этот ген входит в большинство стандартных ГМ-конструкций.
Пятой мишенью является ген рибосом инактивирующего белка I из лаконоса Phytolacca acinosa [9]. Данный ген способен ингибировать развитие вирусов, грибов и бактерий в тканях растений.
Вполне вероятно, что ГМ-конструкции на его основе скоро могут появиться в геномах овощных культур, таких как огурец, перец и томат.
Выделение ДНК для исследования
Выделение ДНК проводилось при помощи усовершенствованных методик (Hong et al., 1995; Foissac et al., 2001).
0,2 г образца было гомогенизировано с 300 мкл экстракционного буфера (0,4 М LiCl, 25mM EDTA, 0,2 M Tris-HCL (pH 9), 2,5% PVP-40) на гомогенизаторе Tissue Lyser II (Qiagen, Germany).
Затем следовала очистка хлороформ/изоамилом 24/1 с последующим улавливанием ДНК при помощи диоксида кремния (силика) с добавлением 96% этанола и 6M NaI.
Далее силику осаждали, промывали 70% спиртом и элюировали ДНК в MQ воду.
Состав ПЦР-смеси и условия амплификации
Все анализы проводились на амплификаторе ПЦР в реальном времени LightCycler 480 II (Roche) с блоком на 96 ячеек. В 10 мкл общей смеси содержалось 2 мкл матрицы (10 нг/мкл ДНК), 1×SYBR®Green PCR Mastermix и 100 нМ каждого праймера.
Программа амплификации: один цикл активации ДНК-полимеразы в течение 10 минут при 95°C, за которым следуют 40 циклов амплификации, 15 секунд при 95°C (стадия денатурации) и 1 минута при 60°C (стадия отжигаудлинения).
Затем проводился анализ температуры плавления полученных продуктов амплификации при постепенном увеличении температуры с 60 до 95°С в течение 20 мин (± 0,6°/20 с).
Полученные данные обрабатывались при помощи программного обеспечения LightCycler® 480 SW 1.5.1.
Разработка маркеров для обнаружения генетически модифицированных культур
При разработке пар праймеров для соответствующих целей применялся унифицированный подход к дизайну праймеров.
Первый шаг состоит в выявлении областей с высокой гомологией последовательностей ДНК в областях nptII, cmvCP, p35S, tNOS и PacPAP из различных последовательностей ГМО ДНК.
Затем для консервативной области (мишени) производился дизайн нескольких пар праймеров, разработанных с использованием программы Primer3Plus с использованием стандартной конфигурации программы.
Анализ на специфичность in silico для каждого праймера проводится путем исследования его последовательности по нескольким общедоступным базам данных ДНК-последовательностей ГМО [10, 11], а также по доступной внутренней информации о последовательностях.
Любой праймер, демонстрирующий гомологию с нерелевантной последовательностью ДНК, исключается из дальнейшего анализа. Оставшиеся праймеры организованы в пары и протестированы экспериментально.
Специфичность анализа
Для оценки корректной работы всех пяти составляющих диагностической системы были произведены, сперва моноплексные ПЦР-реакции, в ходе которых были определены уровни флуоресценции и эффективность реакции для каждой мишени в отдельности.
В качестве матрицы была синтезирована синтетическая цепь ДНК, комплементарная мишени для маркеров. Такой шаг был предпринят в силу того, что ГМ-культуры с ГМ-элементами cmvCP и PacPAP недоступны в нашей стране. После первичного анализа работы пар праймеров был проведен тест на взаимодействие маркерных систем между собой в мультиплексе.
На данный момент не существует алгоритма, который мог бы предсказать степень взаимодействия синтетических олигов друг с другом в одной реакционной смеси. Поэтому маркерные системы для пентаплексной ПЦР-реакции компоновались эмпирически.
Все олигонуклеотиды, приводившие к падению эффективности реакции, подвергались браковке.
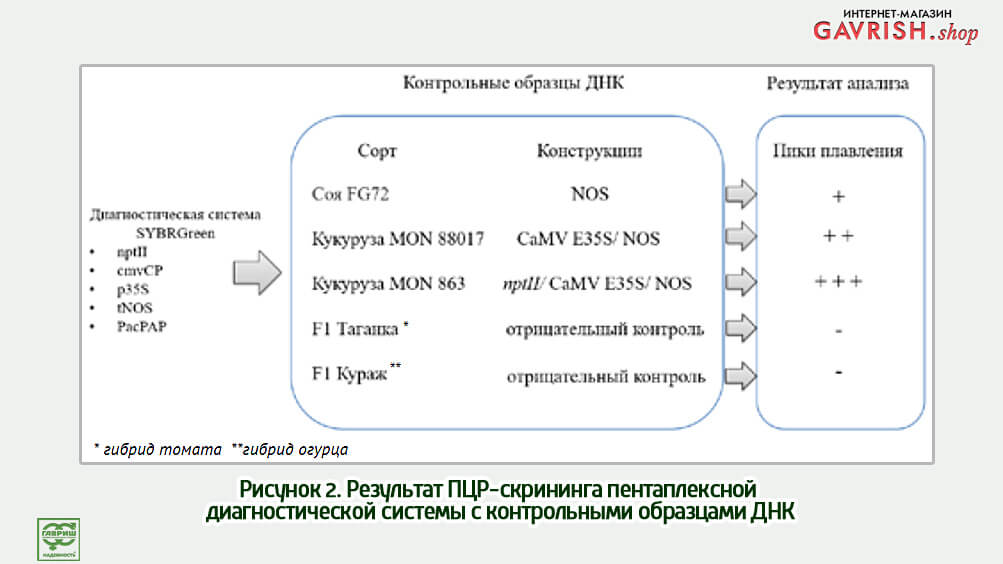
После завершения состава пенталексная диагностическая система была протестирована на ДНК коммерческих образцов разрешенных в нашей стране ГМ-культур.
Подтверждение эффективности тест-системы
В ходе тестирования пентаплексной диагностической системы на искусственно синтезированных матрицах был определен набор из 10 олигонуклеотидов, при котором кинетика их взаимодействия в реакционной смеси не приводила к снижению эффективности анализа.
После подтверждения корректной работы диагностической системы на искусственно синтезированной матрице она была успешно протестирована на коммерческих ГМО-образцах.
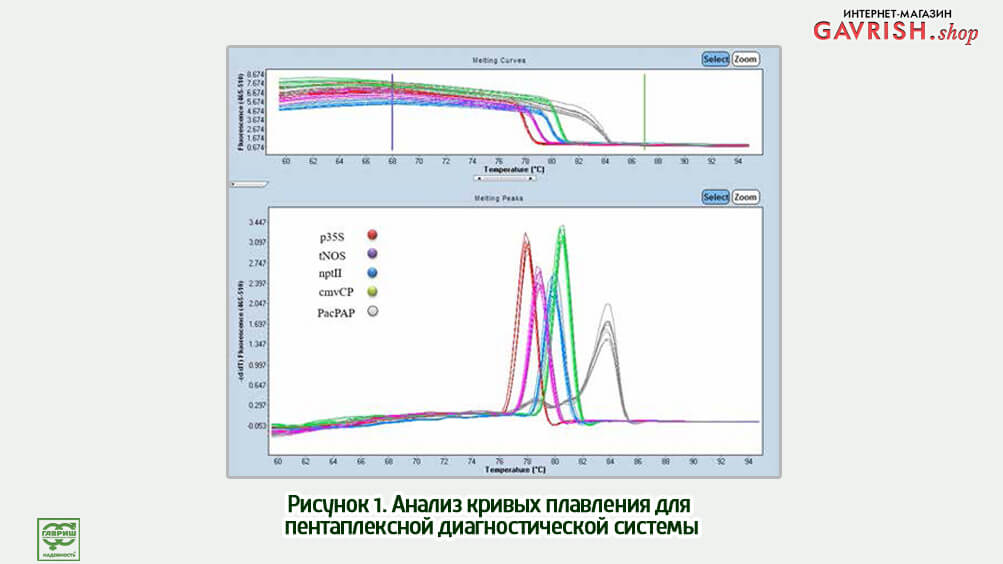
Из приведенного выше результата ПЦР-анализа видно, что диагностическая система эффективно диагностирует nptII, p35S и tNOS конструкции в коммерчески доступных образцах. Причем количество пиков плавления соответствовало ожидаемому количеству мишеней, заявленных в коммерческих ГМ-образцах сои и кукурузы. В то же время в образцах ДНК, которые заведомо не содержали ГМ-конструкций, пики плавления не генерировались.
Выводы
Была разработана новая пентаплексная диагностическая система для проведения рутинного качественного анализа ГМО, основанная на SYBR®Green PCR генетических элементов nptII, cmvCP, p35S, tNOS и PacPAP, которые в настоящее время являются актуальными мишенями при скрининговом анализе ГМО овощных культур.
Данная диагностическая система является надежной в отношении специфичности мишени, так как только мишень-позитивные ДНК-матрицы генерируют экспоненциальную амплификацию.
Анализ кривых плавления сгенерированных ампликонов представляет один пик при ожидаемой температуре. Также данная диагностическая система демонстрирует одну полосу в агарозном геле для каждой мишени, содержащей ГМ-событие, и продукты амплификации соответствуют ожидаемому размеру и предсказанной последовательности ДНК.
Кроме того, данная концепция качественной диагностики, заключающаяся в пентаплексном скрининге при помощи SYBR®Green, является новым подходом в области рутинного анализа ГМО.
Данная система превосходит существующие аналоги как по экономической эффективности и простоте использования, так и по требованиям к оборудованию для real-time PCR.
В данной работе мы продемонстрировали надежность и приемлемость рутинного скрининга ГМО-конструкций на платформе real-time PCR при помощи интерполирующего крестителя SYBR®Green.
Благодаря сертификации поступающих коллекционных образцов при помощи real-time PCR-диагностики селекционно-семеноводческой компанией «Гавриш» ведется надежный контроль над ГМО-продукцией.
За 3 года работы в данном направлении нами не было выявлено ни одного случая попадания ГМ-продукции в коллекцию исходного селекционного материала томата и огурца. Поэтому мы можем гарантировать отсутствие следов ГМО в селекционной работе компании.
---
Будылин М.В., кандидат биологических наук, заместитель генерального директора по молекулярной диагностике и биотехнологии
Тяпкина Д.Ю., научный сотрудник лаборатории молекулярной диагностики
Даниленко А.А., старший лаборант лаборатории молекулярной диагностики
Першин А.Ф., кандидат биологических наук, зав. лаборатории биотехнологии ООО «Селекцентр»
---
Список литературы:
1. C. James, Global status of commercialized biotech/GM crops: 2014, ISAAA Brief 49, 2014.
2. http://www.isaaa.org/gmapprovaldatabase/crop/default.asp?CropID=24&Crop=Tomato
3. Beck, E. et al., 1982. Derived from GeneBank Accession number U0004 19385. Gene 19: 327–336.
4. G. Cottenet, C. Blancpain, V. Sonnard, and P.F. Chuah, (2013) Development and validation of a multiplex real-time PCR method to simultaneously detect 47 targets for the identification of genetically modified organisms, Analytical and Bioanalytical Chemistry, vol. 405, no. 21, pp. 6831–6844.
5. Yenofsky R.L., Fine M., Pellow J.W, (1990) A mutant neomycin phosphotransferase II gene reduces the resistance of transform ants to antibiotic selection pressure. Proc Nat! Acad Sci USA 87: 3435–3439.
6. Feng J., Liang Y., Wang F., Chen J. (2013) Detection of genetically modified tomato using PCR coupled with muParaflo microfluidics microarrays. J. Nanosci Nanotechnol; 13(12):8266-74.
7. Odell J.T., Nagy F., Chua N-H. Identification of DNA sequences required for activity of the cauliflower mosaic virus 35S promoter. Nature. 1985; 313: 810–2. Available at doi:10.1038/313810a0.
8. Bruderer S., Leitner K.E., Lindenmeyer J., (2003) Genetically modified (GM) crops: molecular and regulatory details, BATS, centre for biosafety and sustainability, version 2. Available at http://www.bats.ch/gmo-watch/index.php.
9. Cao B., Lei J., Chen G., Cao P., Liu X., Chen Q., and Wei X., Testing of disease-resistance of pokeweed antiviral protein gene (PacPAP) in transgenic cucumber (Cucumis sativus), Afr. J. Biotechnol., 2011, vol. 10, no. 36, pp. 6883–6890.
10. EMBL database: http://www.ebi.ac.uk/embl/
11. RefSeq NCBI database: http://www.ncbi.nlm.nih.gov/RefSeq/













